packages_to_install <- c("RJDemetra", "remotes", "rjdqa", "ggdemetra", "rjdmarkdown", "rjwsacruncher")
packages <- packages_to_install[! packages_to_install %in% installed.packages()[,"Package"]]
if (length(packages) > 0) {
install.packages(packages)
}
packages_to_install_git <- c("rjd3toolkit", "rjd3x13", "rjd3tramoseats", "rjd3providers", "rjdemetra3")
packages_git <- packages_to_install_git[! packages_to_install_git %in% installed.packages()[,"Package"]]
if (length(packages_git) > 0) {
# # Configurer si besoin le proxy
# proxy <- "proxy_a_definir"
# Sys.setenv(HTTPS_PROXY = proxy)
remotes::install_github(
sprintf("rjdemetra/%s", packages_git),
# option utile dans certaines installations portables de Java :
INSTALL_opts = "--no-multiarch")
}
if (! "rjdworkspace" %in% installed.packages()[,"Package"])
remotes::install_github("InseeFrLab/rjdworkspace",
INSTALL_opts = "--no-multiarch")5 - JDemetra+ en production
Désaisonnaliser une série temporelle
L’objectif de ce TP est d’apprendre à manipuler des workspaces pour une mise en production.
Pour installer tous les packages utiles de ce TP, lancer le programme :
Lors de la mise en production, le plus simple est de manipuler des workspaces et de mettre à jour les modèles, lors de l’arrivée de nouvelles données à travers le JWSACruncher. Pour faciliter son utilisation depuis R, le package rjwsacruncher peut être utilisé.
Lorsque les workspaces sont créés depuis R, on perd toutes les métadonnées (lien vers les fichiers, commentaires, etc.), une solution pour cela : utiliser rjdworkspace (package non publié sur le CRAN) pour récupérer ces données depuis un autre workspace. Pour la version 3, rjdemetra3 contient toutes les fonctionnalités de rjdworkspace.
Dans ce TP on utilisera les données du package RJDemetra mais n’hésitez pas à utiliser vos propres séries.
1 JWSACruncher et rjwsacruncher
1.1 Configuration du JWSACruncher
Le JWSACruncher est téléchargeable ici : https://github.com/jdemetra/jwsacruncher/releases.
Pour utiliser les dernières versions il faut avoir une version de Java supérieure à la 8, si ce n’est pas le cas, il faut télécharger une version portable de Java et configurer le JWSACruncher en conséquence (voir manuel d’installation). Cette opération n’est pas utile si l’on utilise une version 3.x.y de JDemetra+ (une version portable de Java est “embarquée” avec le JWSACruncher). Ces manipulations peuvent aussi se faire à partir de rjwsacruncher :
# install.packages("rjwsacruncher") # Si pas déjà installé
library(rjwsacruncher)
# Télécharge l'archive du JWSACruncher et la met sur le D:/
download_cruncher(
"D:/",
# Par défaut on télécharge la dernière version du cruncher en V2
v3 = FALSE)
# Dézipper l'archive configurer avec une version portable de Java :
jwsacruncher_path <- "D:/jwsacruncher-2.2.4-bin/bin/jwsacruncher.bat"
# Lien vers le fichier java.exe de la version portable de Java :
java_path <- "D:/Java8/bin/java.exe"
configure_jwsacruncher(jwsacruncher_path, java_path)Pour indiquer à rjwsacruncher où se trouve le JWSACruncher, le plus simple est de mettre à jour l’option cruncher_bin_directory :
# Chemin vers le dossier bin du JWSACruncher
# Remplacer "jwsacruncher-2.2.4/bin" par votre propre chemin.
options(cruncher_bin_directory =
"jwsacruncher-2.2.4/bin")
getOption("cruncher_bin_directory") # Pour afficher la valeur actuelle[1] "jwsacruncher-2.2.4/bin"1.2 Utilisation du JWSACruncher
Pour éviter que le package rjwsacruncher soit trop volumineux, il ne contient pas le JWSAcruncher de JDemetra+. Ce dernier peut être téléchargé à l’adresse suivante : https://github.com/jdemetra/jdemetra-app/releases ou en utilisant la fonction rjwsacruncher::download_cruncher(). Pour sa configuration avec une version portable, voir le manuel d’installation.
Pour lancer le JWSACruncher il faut trois fichiers :
- un fichier contenant les paramètres sur la méthode de rafraîchissement à utilisée pour mettre à jour le workspace (créé à partir de la fonction
create_param_file()oulist2param_file()) ;
- un workspace valide de JDemetra+ ;
- l’adresse vers le JWSACruncher (option
cruncher_bin_directory).
Dans le package rjwsacruncher, les principales fonctions associées au lancement du JWSACruncher sont :
create_param_file()oulist2param_file()qui permet de créer le fichier de paramètres ;
cruncher()qui permet de lancer le JWSACruncher sur un workspace à partir d’un fichier de paramètres ;
cruncher_and_param()qui permet de lancer le JWSACruncher tout en créant le fichier de paramètres et de personnaliser certaines sorties du JWSACruncher.
1.2.1 Création du fichier de paramètres avec create_param_file()
Les paramètres de la fonction create_param_file() sont les mêmes que ceux décrits dans le wiki du JWSACruncher de JDemetra+ (https://github.com/jdemetra/jwsacruncher/wiki). Les trois paramètres les plus importants de create_param_file() sont :
policyqui est la méthode de rafraîchissement utilisée (voir tableau ci-dessous).
| Option sous JDemetra+ | Option du cruncher | Signification |
|---|---|---|
| Partial concurrent adjustment -> Fixed model | current | Le modèle ARIMA, les outliers et les autres paramètres du modèle de régression ne sont ni ré-identifiés ni ré-estimés. Le schéma de décomposition est inchangé. |
| Partial concurrent adjustment -> Estimate regression coefficients | fixedparameters (ou fixed) | Le modèle ARIMA, les outliers et les autres paramètres du modèle regARIMA ne sont pas ré-identifiés. Les coefficients du modèle ARIMA sont fixés et les autres paramètres du modèle de régression sont ré-estimés. Le schéma de décomposition est inchangé. |
| Partial concurrent adjustment -> Estimate regression coefficients + Arima parameters | parameters (paramètre par défaut) | Le modèle ARIMA, les outliers et les autres paramètres du modèle de régression ne sont pas ré-identifiés mais sont tous ré-estimés. Le schéma de décomposition est inchangé. |
| Partial concurrent adjustment -> Estimate regression coefficients + Last outliers | lastoutliers | Le modèle ARIMA, les outliers (sauf ceux de la dernière année) et les autres paramètres du modèle de régression ne sont pas ré-identifiés mais sont tous ré-estimés. Les outliers de la dernière année sont ré-identifiés. Le schéma de décomposition est inchangé. |
| Partial concurrent adjustment -> Estimate regression coefficients + all outliers | outliers | Le modèle ARIMA et les paramètres du modèle regARIMA autres que les outliers ne sont pas ré-identifiés mais ré-estimés. Tous les outliers sont ré-identifiés. Le schéma de décomposition est inchangé. |
| Partial concurrent adjustment -> Estimate regression coefficients + Arima model | stochastic | Ré-identification de tous les paramètres du modèle regARIMA hormis les variables calendaires. Le schéma de décomposition est inchangé. |
| Concurrent | complete ou concurrent | Ré-identification de tout le modèle regARIMA. |
matrix_itemqui est une chaîne de caractères contenant les noms des paramètres à exporter. Par défaut, ce sont ceux de l’optiondefault_matrix_item. On peut donc au choix modifier l’optiondefault_matrix_itemou le paramètrematrix_item:
library(rjwsacruncher)
# Pour afficher les paramètres par défaut :
getOption("default_matrix_item")
# Pour modifier les paramètres par défaut pour n'exporter par exemple
# que les critères d'information :
options(default_matrix_item = c("likelihood.aic",
"likelihood.aicc",
"likelihood.bic",
"likelihood.bicc"))tsmatrix_seriesqui est une chaîne de caractères contenant les noms des paramètres à exporter. Par défaut, ce sont ceux de l’optiondefault_tsmatrix_series. On peut donc au choix modifier l’optiondefault_tsmatrix_seriesou le paramètretsmatrix_series:
# Pour afficher les paramètres par défaut :
getOption("default_tsmatrix_series")
# Pour modifier les paramètres par défaut pour n'exporter par exemple que
# la série désaisonnalisée et ses prévisions :
options(default_tsmatrix_series = c("sa", "sa_f"))Pour voir l’ensemble des paramètres, il suffit d’utiliser sous R la commande ?create_param_file.
Après cela, il ne reste plus qu’à créer le fichier de paramètres. Le fichier de paramètre est différent entre la version 3.0.0 de JDemetra+ et les versions antérieures. Les fonctions de création des fichiers de paramètres ont un paramètre v3 qui permet de spécifier si l’on veut que le fichier soit compatible avec la version 3.0.0. Sa valeur par défaut est getOption("is_cruncher_v3") : si l’on utilise la version 3.0.0 ou plus, le plus simple est donc de changer cette option options(is_cruncher_v3 = TRUE). Ci-dessous quelques exemples.
# Un fichier parametres.param sera créé sous D:/
# avec la politique de rafraîchissement "lastoutliers"
# et les autres paramètres par défaut
create_param_file(dir_file_param = "D:/",
policy = "lastoutliers")
# Si l'on a modifié les options "default_matrix_item" et "default_tsmatrix_series" pour
# n'exporter que les critères d'information, la série désaisonnalisée et ses
# prévisions, la commande précédente est équivalent à :
create_param_file(dir_file_param = "D:/",
policy = "lastoutliers",
matrix_item = c("likelihood.aic", "likelihood.aicc",
"likelihood.bic", "likelihood.bicc"),
tsmatrix_series = c("sa", "sa_f"))Utiliser la fonction create_param_file() pour créé un fichier de paramètres permettant de mettre à jour un workspace :
- En reestimant le modèle ARIMA, les outliers et les autres paramètres du modèle de régression et en re-identifiant les outliers uniquement sur la dernière année.
- En exportant la statistique M7, la statistique Q-M2 et les tests de jours ouvrables résiduels ;
- En exportant La série brute, la série désaisonnalisée et la tendance (de manière verticale).
Pour voir les noms des paramètres, utiliser la fonction default_param_file() ou bien aller sur la page https://github.com/jdemetra/jwsacruncher/wiki/Output-dictionaries.
dir <- tempdir()
create_param_file(
dir_file_param = dir,
policy = "lastoutliers",
matrix_item = c(
"m-statistics.m7",
"m-statistics.q-m2",
"diagnostics.residual trading days tests.f-test on sa (td):2",
"diagnostics.residual trading days tests.f-test on i (td):2"
),
tsmatrix_series = c("y", "sa", "t"),
csv_layout = "vtable"
)Les fichiers de paramètres peuvent être lus avec read_param_file() qui renvoie une liste qui peut être modifiée et exportée avec list2param_file() :
param_f <- read_param_file(file.path(dir, "parameters.param"))
str(param_f)List of 7
$ config :List of 4
..$ bundle : chr "10000"
..$ csv_layout : chr "vtable"
..$ csv_separator: chr ";"
..$ ndecs : chr "6"
$ policy : chr "lastoutliers"
$ refreshall : logi TRUE
$ output : NULL
$ matrix_item : chr [1:4] "m-statistics.m7" "m-statistics.q-m2" "diagnostics.residual trading days tests.f-test on sa (td):2" "diagnostics.residual trading days tests.f-test on i (td):2"
$ tsmatrix_series: chr [1:3] "y" "sa" "t"
$ paths_path : NULLLes fichiers de paramètres par défaut peuvent être obtenus avec la fonction default_param_file().
1.2.2 Lancement du JWSACruncher
Pour lancer le JWSACruncher avec cruncher() ou cruncher_and_param(), il faut spécifier le chemin d’accès au dossier contenant le JWSACruncher (paramètre cruncher_bin_directory) ainsi que celui du workspace à traiter (paramètre workspace). Si cela a déjà été fait dans la section 1.1, ne pas prendre en compte le paragraphe suivant
Par défaut, le chemin d’accès au dossier du JWSACruncher est celui contenu dans le paramètre cruncher_bin_directory : il suffit donc de modifier une seule fois cette option afin qu’elle s’applique à toutes les exécutions du JWSACruncher. Le chemin à indiquer est celui du dossier contenant le fichier jwsacruncher.bat, situé dans le dossier “Bin” du dossier d’installation du JWSACruncher. Ainsi, s’il a été installé sous D:\jdemetra-cli-2.2.4, le fichier jwsacruncher.bat sera présent sous D:\jdemetra-cli-2.2.4\bin. Il faut donc modifier l’option cruncher_bin_directory de la façon suivante :
options(cruncher_bin_directory = "D:/jdemetra-cli-2.2.4/bin/")Si aucun chemin de workspace n’est renseigné, une fenêtre s’ouvre, invitant à sélectionner le workspace sur lequel on souhaite lancer le JWSACruncher.
# Remplacer ici "workspace.xml" par le chemin vers votre workspace
cruncher(workspace = "workspace.xml",
# Remplacer ici "parameters.param" par le chemin vers votre fichier de paramètres
param_file_path = "parameters.param"
)Si vous n’avez pas de workspace vous pouvez utiliser le code suivant pour en générer un :
library(RJDemetra)
spec_x13 <- x13_spec(spec = "RSA5c")
jws <- new_workspace()
new_multiprocessing(jws, "sa1")
for (nom_series in colnames(ipi_c_eu)){
model <- jx13(ipi_c_eu[,nom_series], spec_x13)
add_sa_item(jws, "sa1", model, nom_series)
}
save_workspace(jws, "workspace.xml")Si non spécifié dans le fichier des paramètres, les résultats sont exportés dans le sous dossier "Output" du workspace (pour le workspace.xml, les résultats seront donc sous workspace/Output/). On peut aussi créer le fichier des paramètres et lancer le JWSAcruncher avec la fonction cruncher_and_param. Cette fonction permet aussi de renommer les dossiers exportées avec les noms des multi-processings utilisés dans JDemetra+ (évite d’avoir des dossiers du type SAProcessing-1)1.
cruncher_and_param(
workspace = "workspace.xml",
policy = "lastoutliers",
matrix_item = c(
"m-statistics.m7",
"m-statistics.q-m2",
"diagnostics.residual trading days tests.f-test on sa (td):2",
"diagnostics.residual trading days tests.f-test on i (td):2"
),
tsmatrix_series = c("y", "sa", "t"),
csv_layout = "vtable"
)2 Mise à jour des données avec rjdemetra3
Avec les packages de rjdemetra3 vous pouvez également directement rafraîchir les spécifications depuis R, la chaîne de production peut donc être entièrement codée en R ! Pour cela il faut utiliser la fonction rjd3x13::x13_refresh() :
y <- ipi_c_eu[,"FR"]
y_est <- window(y, end = 2019)
mod <- rjd3x13::x13(y_est)
mod$result$preprocessingLog-transformation: yes
SARIMA model: (2,1,1) (0,1,1)
Coefficients
Estimate Std. Error T-stat
phi(1) -0.01262 0.10670 -0.118
phi(2) 0.15483 0.07314 2.117
theta(1) -0.53470 0.09811 -5.450
btheta(1) -0.69469 0.04642 -14.965
Regression model:
Estimate Std. Error T-stat
td 0.0069229 0.0002921 23.703
easter -0.0225158 0.0041277 -5.455
LS (2008-11-01) -0.0855201 0.0168275 -5.082
LS (2009-01-01) -0.0720329 0.0168047 -4.286
AO (2011-05-01) 0.1292294 0.0167144 7.732
Number of observations: 349
Number of effective observations: 336
Number of parameters: 10
Loglikelihood: 827.5883
Adjusted loglikelihood: -724.056
Standard error of the regression (ML estimate): 0.02035457
AIC: 1468.112
AICC: 1468.789
BIC: 1506.283 spec_x13_ref <- rjd3x13::x13_refresh(
# Point spec: la specification de laquelle on part
spec = mod$result_spec,
# Domain spec: Specification utilisée pour l'estimation
# permet de définir les contraintes (CJO, valeurs possibles ARIMA...)
refspec = mod$estimation_spec,
# Politique de rafraîchissement
policy = "Outliers",
# Période où les outliers sont figés :
period = 12,
start = start(y_est),
end = end(y_est) - c(1, 0)
)
mod_ref <- rjd3x13::x13(y, spec_x13_ref)
mod_ref$result$preprocessingLog-transformation: yes
SARIMA model: (2,1,1) (0,1,1)
Coefficients
Estimate Std. Error T-stat
phi(1) -0.05032 0.11064 -0.455
phi(2) 0.12922 0.07407 1.745
theta(1) -0.56250 0.10270 -5.477
btheta(1) -0.70607 0.04405 -16.030
Regression model:
Estimate Std. Error T-stat
td 0.0070469 0.0002873 24.529
easter -0.0226977 0.0041604 -5.456
LS (2008-11-01) -0.0852825 0.0174225 -4.895
LS (2009-01-01) -0.0716876 0.0173995 -4.120
AO (2011-05-01) 0.1290416 0.0171996 7.503
TC (2020-03-01) -0.2046281 0.0202037 -10.128
TC (2020-04-01) -0.2804802 0.0203271 -13.798
Number of observations: 372
Number of effective observations: 359
Number of parameters: 12
Loglikelihood: 875.4961
Adjusted loglikelihood: -781.4164
Standard error of the regression (ML estimate): 0.02086258
AIC: 1586.833
AICC: 1587.735
BIC: 1633.433 # Le modèle est différent de celui obtenu en réestimant
# avec la spécification par défaut
rjd3x13::x13(y)$result$preprocessingLog-transformation: no
SARIMA model: (2,1,1) (0,1,1)
Coefficients
Estimate Std. Error T-stat
phi(1) 0.05291 0.10635 0.497
phi(2) 0.18672 0.07339 2.544
theta(1) -0.52138 0.10129 -5.147
btheta(1) -0.66132 0.05065 -13.056
Regression model:
Estimate Std. Error T-stat
td 0.69265 0.03143 22.039
lp 2.09031 0.69411 3.012
easter -2.54757 0.44179 -5.766
LS (2008-11-01) -9.27443 1.75769 -5.276
LS (2009-01-01) -7.28376 1.75607 -4.148
AO (2011-05-01) 13.18697 1.80957 7.287
AO (2020-03-01) -21.14917 2.12203 -9.966
TC (2020-04-01) -35.64811 2.09187 -17.041
Number of observations: 372
Number of effective observations: 359
Number of parameters: 13
Loglikelihood: -795.0695
Standard error of the regression (ML estimate): 2.193228
AIC: 1616.139
AICC: 1617.194
BIC: 1666.622 3 Mise à jour des metadonnées avec rjdworkspace
Lorsque l’on manipule des objets depuis RJDemetra, plusieurs informations sont perdues par rapport à JDemetra+, dont :
le lien vers les données d’origine
les éventuels commentaires que l’on peut faire
Toutes ces informations sont les metadata. Lorsque vous créer un workspace depuis JDemetra+, vous pouvez par exemple voir ces données en ouvrant le fichier SAProcessing/SAProcessing-1.xml associé au dossier de votre workspace. Dans l’exemple ci-dessous, dans les premières lignes de ce fichier Excel on peut voir les données utilisées, le nom de la série et dans la partie “metaData” le chemin vers le fichier Excel contenant les données :
<item name="sa1">
<subset>
<item name="ts">
<ts name="IPI BE">
<freq>12</freq>
<firstYear>2000</firstYear>
<firstPeriod>1</firstPeriod>
<data>...</data>
<metaData>
<property name="@timestamp" value="Sun Oct 08 17:19:31 CEST 2023"/>
<property name="@source" value="XCLPRVDR"/>
<property name="@id" value="demetra://tsprovider/XCLPRVDR/20111201/SERIES?file=%2FUsers%2Falainquartierlatente%2FDesktop%2Fdata.xlsx#seriesName=BE&sheetName=IPI"/>
</metaData>
</ts>
</item>
....Supposons que ce soit le workspace utilisé en production (i.e. : le workspace sur lequel vous lancez rjwsacruncher). On va se placer dans le cas où l’on souhaite modifier ce workspace depuis R. On repartira du workspace ws_prod.xml qui est dans le .zip disponible sous /data/data_prod.zip. Pour le télécharger depuis R :
dir_dl <- "."
download.file("https://AQLT.github.io/formation.2023.cvs/data/data_prod.zip",
file.path(dir_dl, "data_prod.zip"))
unzip(file.path(dir_dl, "data_prod.zip"),
exdir = dir_dl)Créer un nouveau workspace ws_prod_tmp.xml où l’on a modifié toutes les spécifications de votre workspace en rajoutant un AO en janvier 2020.
library(RJDemetra)
# Chargement du workspace
jws <- load_workspace(file.path(dir_dl, "ws_prod.xml"))
compute(jws)
# Import de tous les modèles
# On a une liste qui contient autant d'éléments que de multiprocessings
# et chaque élément est une liste qui contient autant d'éléments que de modèle
# dans le multiprocessing considéré
all_models <- get_model(jws, progress_bar = FALSE)
jws2 <- new_workspace()
for(sa_name in names(all_models)){
new_multiprocessing(jws2, sa_name)
for (series_name in names(all_models[[sa_name]])){
new_spec <- x13_spec(all_models[[sa_name]][[series_name]],
usrdef.outliersEnabled = TRUE,
usrdef.outliersType = c("AO"),
usrdef.outliersDate = c("2020-01-01"))
new_jmod <- jx13(get_ts(all_models[[sa_name]][[series_name]]),
new_spec)
add_sa_item(workspace = jws2, multiprocessing = sa_name,
sa_obj = new_jmod,
name = series_name)
}
}
save_workspace(jws2, file.path(dir_dl, "ws_prod_tmp.xml"))Si vous ouvrez maintenant le fichier ws_prod_tmp/SAProcessing/SAProcessing-1.xml vous remarquez donc qu’il n’y a plus les parties metaData !
Pour les mettre à jour il existe deux fonctions dans rjdworkspace :
update_medataqui, à partir d’un workspace de référence, met à jour un workspace (ws_prod_tmp.xml) en faisant un matching sur le nom des séries (il y a donc potentiellement un problème si on a plusieurs séries avec le même nom)update_metadata_roughly(), à partir d’un workspace de référence, met à jour un workspace (ws_prod_tmp.xml) en fonction de l’ordre de la série dans le modèle (le premier modèle dews_prod_tmp.xmlest mis à jour avec les informations du premier modèle de l’autre workspace, etc.).
Dans notre cas, update_metadata_roughly() suffit :
library(rjdworkspace)
jws3 <- update_metadata_roughly(
jws, # D'abord le workspace qui contient les metadata
jws2 # Ensuite le workspace à mettre à jour
)
# Il reste à sauvegarder le nouveau workspace
save_workspace(jws3, file.path(dir_dl, "ws_prod_tmp2.xml"))4 Mise à jour des metadonnées avec rjdemetra3
Dans rjdemetra3 il existe différentes fonctions pour modifier les métadonnées des SAItems : rjdemetra3::set_comment(), rjdemetra3::set_context(), rjdemetra3::set_domain_specification(), rjdemetra3::set_name(), rjdemetra3::set_priority(), rjdemetra3::set_raw_data(), rjdemetra3::set_specification(), rjdemetra3::set_ts_metadata(), rjdemetra3::put_ts_metadata(), rjdemetra3::set_ts(). Le package rjd3providers permet également, grâce à rjdemetra3, de mettre à jour les providers depuis R (i.e. : les chemins vers les données).
Les informations sur les metadata associés à la série temporelle peut être lue en utilisant la fonction get_ts() :
jws_prod <- rjdemetra3::.jws_load(file.path(dir_dl, "ws_prod.xml"))
jws_prod_tmp <- rjdemetra3::.jws_load(file.path(dir_dl, "ws_prod_tmp.xml"))
jsa1 <- rjdemetra3::.jsap_sa(rjdemetra3::.jws_sap(jws_prod, 1), 1)
jsa1_tmp <- rjdemetra3::.jsap_sa(rjdemetra3::.jws_sap(jws_prod_tmp, 1), 1)
rjdemetra3::get_ts(jsa1)[1:3] # 4e élément est la série brute $name
[1] "IPI\nBE"
$moniker
$source
[1] ""
$id
[1] "d484d031-64cc-45b3-aff8-66b280963958"
attr(,"class")
[1] "JD3_TSMONIKER"
$metadata
$metadata$`@timestamp`
[1] "Sun Oct 08 17:19:31 CEST 2023"
$metadata$`@source`
[1] "XCLPRVDR"
$metadata$`@id`
[1] "demetra://tsprovider/XCLPRVDR/20111201/SERIES?file=%2FUsers%2Falainquartierlatente%2FDesktop%2Fdata.xlsx#seriesName=BE&sheetName=IPI"rjdemetra3::get_ts(jsa1_tmp)[1:3]$name
[1] "BE"
$moniker
$source
[1] ""
$id
[1] "35e3351b-9232-48a7-a8a8-fdaee23224f9"
attr(,"class")
[1] "JD3_TSMONIKER"
$metadata
NULLEn utilisant la fonction rjdemetra3::set_ts_metadata(), reproduire la fonction rjdworkspace::update_metadata_roughly(). Il faut pour cela parcourir tous les SAProcessing et tous les SAItems avec les fonctions vues dans les précédents TP. Vérifier le résultat.
update_metadata_roughly2 <- function(jws1, jws2) {
for (i_mp in seq_len(rjdemetra3:::.jws_sap_count(jws1))) {
jmp_ws1 <- rjdemetra3::.jws_sap(jws1, i_mp)
jmp_ws2 <- rjdemetra3::.jws_sap(jws2, i_mp)
for (i_sa in seq_len(rjdemetra3::.jsap_sa_count(jmp_ws1))) {
rjdemetra3::set_ts_metadata(jmp_ws2, i_sa,
rjdemetra3::.jsap_sa(jmp_ws1, i_sa))
}
}
return(invisible(TRUE))
}
update_metadata_roughly2(jws_prod,
jws_prod_tmp)
jsa1_tmp <- rjdemetra3::.jsap_sa(rjdemetra3::.jws_sap(jws_prod_tmp, 1), 1)
rjdemetra3::get_ts(jsa1_tmp)[1:3]$name
[1] "BE"
$moniker
$source
[1] ""
$id
[1] "d484d031-64cc-45b3-aff8-66b280963958"
attr(,"class")
[1] "JD3_TSMONIKER"
$metadata
$metadata$`@timestamp`
[1] "Sun Oct 08 17:19:31 CEST 2023"
$metadata$`@source`
[1] "XCLPRVDR"
$metadata$`@id`
[1] "demetra://tsprovider/XCLPRVDR/20111201/SERIES?file=%2FUsers%2Falainquartierlatente%2FDesktop%2Fdata.xlsx#seriesName=BE&sheetName=IPI"Comme vous le remarquez, le chemin absolu vers les données est inscrit dans le workspace. Si vous lancer le JWSACruncher sur ce workspace vous devriez avoir une erreur puisque le fichier n’est pas au même endroit chez vous ! Ce fichier est dans l’archive data_prod.zip. Le chemin peut également être mis à jour grâce à rjd3providers :
jsap <- rjdemetra3::.jws_sap(jws_prod_tmp, 1)
jsa <- rjdemetra3::.jsap_sa(jsap, 1)
# Il faut changer le paramètre @id
rjdemetra3::get_ts(jsa)$metadata$`@timestamp`
[1] "Sun Oct 08 17:19:31 CEST 2023"
$`@source`
[1] "XCLPRVDR"
$`@id`
[1] "demetra://tsprovider/XCLPRVDR/20111201/SERIES?file=%2FUsers%2Falainquartierlatente%2FDesktop%2Fdata.xlsx#seriesName=BE&sheetName=IPI"# On extrait l'objet java
jsa_id <- rjdemetra3::.jsa_ts_metadata(jsa, "@id")
full_path_f <- base::normalizePath(file.path(dir_dl, "data_ws_prod.xlsx"))
nid <- rjd3providers::spreadsheet_change_file(jsa_id,
full_path_f)
rjdemetra3::put_ts_metadata(jsap, 1, "@id", nid)
jsa <- rjdemetra3::.jsap_sa(jsap, 1)
rjdemetra3::get_ts(jsa)$metadata$`@timestamp`
[1] "Sun Oct 08 17:19:31 CEST 2023"
$`@id`
[1] "demetra://tsprovider/XCLPRVDR/20111201/SERIES?file=%2FUsers%2Falainquartierlatente%2FDesktop%2FLogiciels%2Fformations%2Fformation.2023.cvs%2FTP%2Fdata_ws_prod.xlsx#seriesName=BE&sheetName=IPI"
$`@source`
[1] "XCLPRVDR"On peut aussi passer d’un fichier Excel à un fichier CSV :
jsap <- rjdemetra3::.jws_sap(jws_prod_tmp, 1)
i_serie <- 2
jsa <- rjdemetra3::.jsap_sa(jsap, i_serie)
name_series <- rjdemetra3::.jsa_name(jsa)
name_series[1] "BG"# On importe le fichier CSV :
full_path_f <- base::normalizePath(file.path(dir_dl, "data_ws_prod.csv"))
txt_data = rjd3providers::txt_series(
full_path_f, series = i_serie, delimiter = "SEMICOLON",
gathering.includeMissing = FALSE,
txtQualifier = "DOUBLE_QUOTE", gathering.period = 12
)
rjdemetra3::get_ts(jsa)$metadata$`@timestamp`
[1] "Sun Oct 08 17:19:31 CEST 2023"
$`@source`
[1] "XCLPRVDR"
$`@id`
[1] "demetra://tsprovider/XCLPRVDR/20111201/SERIES?file=%2FUsers%2Falainquartierlatente%2FDesktop%2Fdata.xlsx#seriesName=BG&sheetName=IPI"rjdemetra3::put_ts_metadata(jsap, i_serie, "@source", txt_data$moniker$source)
rjdemetra3::put_ts_metadata(jsap, i_serie, "@id", txt_data$moniker$id)
jsa <- rjdemetra3::.jsap_sa(jsap, 2)
rjdemetra3::get_ts(jsa)$metadata$`@timestamp`
[1] "Sun Oct 08 17:19:31 CEST 2023"
$`@id`
[1] "demetra://tsprovider/Txt/20111201/SERIES?datePattern=&delimiter=SEMICOLON&file=%2FUsers%2Falainquartierlatente%2FDesktop%2FLogiciels%2Fformations%2Fformation.2023.cvs%2FTP%2Fdata_ws_prod.csv&frequency=Monthly&ignoreNumberGrouping=true&locale=&textQualifier=DOUBLE_QUOTE#seriesIndex=1"
$`@source`
[1] "Txt"5 Améliorer le suivi de la production
Lors de la production courante, on a généralement peu de temps pour étudier tous les modèles de désaisonnalisation. peut grandement vous aider, en faisant des graphiques automatiques, des rapports, des tableaux de bord…
Plusieurs packages peuvent vous aider à faire cela dont :
ggdemetra pour créer facilement des graphiques ggplot2 à partir des modèles de RJDemetra ;
rjdqa pour créer des tableaux de bord ;
rjdmarkdown pour un meilleur rendu avec rmarkdown des modèles de RJDemetra.
Pour les packages liés à la version 3 :
rjd3report : devrait rassembler
rjdqaetrjdmarkdown(pour l’instant il n’y a querjd3report::simple_dashboard()).
5.1 ggdemetra
ggdemetra permet de faire de la désaisonnalisation comme une couche supplémentaire de ggplot2.
geom_sa(): pour ajouter une série temporelle associée à la désaisonnalisation (tendance, série désaisonnalisée, etc.) ;geom_outlier(): pour ajouter les points atypiques corrigés dans le pre-ajustement ;geom_arima(): pour ajouter le modèle ARIMA ;geom_diagnostics(): pour ajouter un tableau avec des diagnostics.
Par exemple, pour tracer les séries désaisonnalisées d’un ensemble de séries temporelles :
library(ggdemetra)
dataGraph <- ts2df(na.omit(ipi_c_eu[,c("FR", "UK", "IT")]))
dataGraph <- reshape2::melt(dataGraph, id = "date")
ggplot(data = dataGraph, aes (x = date, y = value, color = variable)) +
geom_sa(component = "sa", frequency = 12, spec = x13_spec("RSA4c"))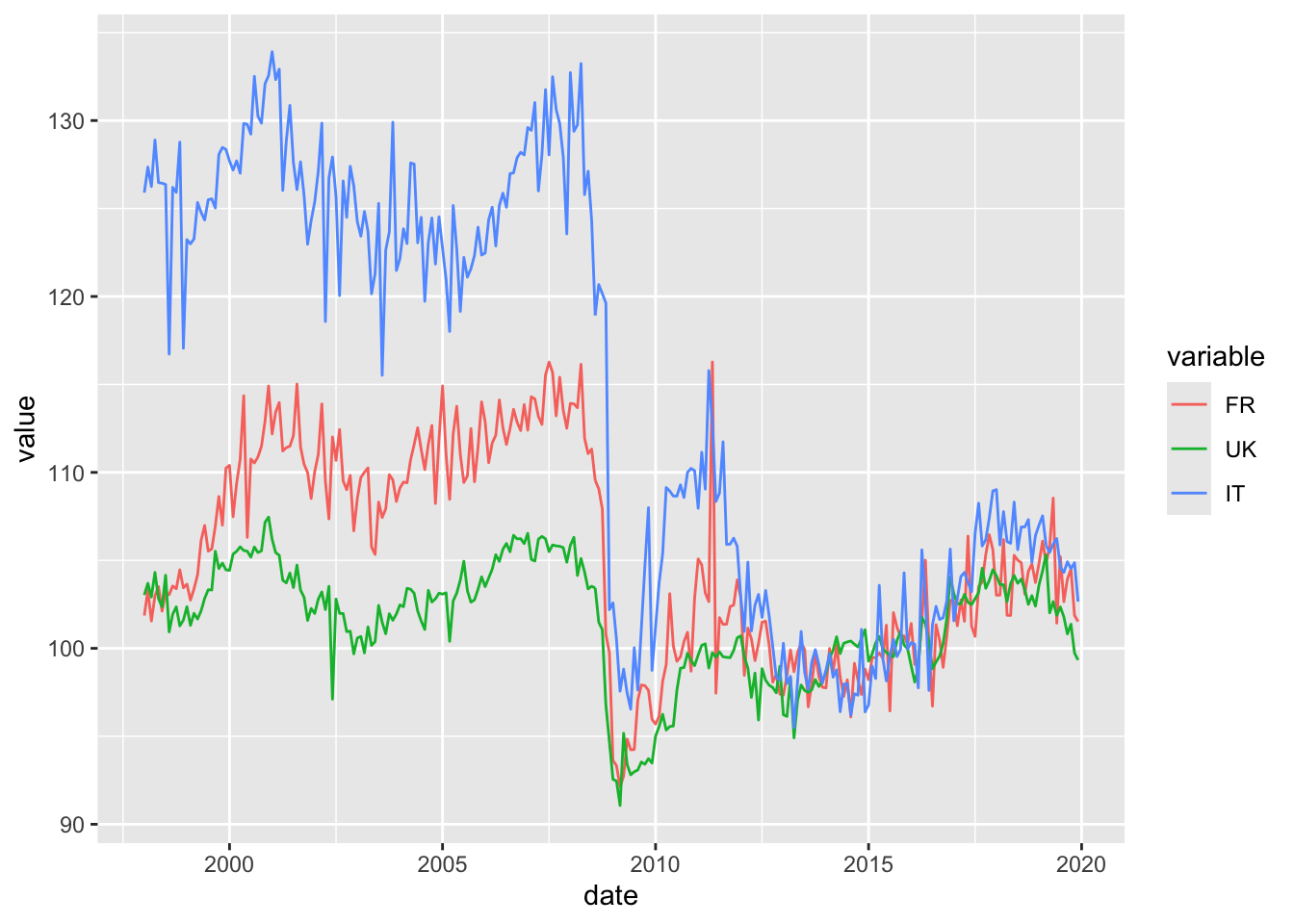
ggplot2 grâce à ggdemetra.
Si vous avez déjà estimé un modèle avec RJDemetra, vous pouvez directement initialiser votre graphique grâce à la fonction init_ggplot :
mod <- x13(window(ipi_c_eu[,"FR"], end = 2019))
diagnostics <- c(`Combined test` = "diagnostics.combined.all.summary",
`Residual qs-test (p-value)` = "diagnostics.qs",
`Residual f-test (p-value)` = "diagnostics.ftest")
p_sa <- init_ggplot(mod) +
geom_line(color = "#F0B400") +
geom_sa(component = "y_f", linetype = 2,
color = "#F0B400") +
geom_sa(component = "sa", color = "#155692") +
geom_sa(component = "sa_f", color = "#155692", linetype = 2) +
geom_outlier(geom = "label_repel",
coefficients = TRUE,
ylim = c(NA, 65),
arrow = arrow(length = unit(0.03, "npc"),
type = "closed", ends = "last"),
digits = 2) +
geom_arima(geom = "label",
x_arima = -Inf, y_arima = Inf,
vjust = 1.2, hjust = -0.1)
p_sa +
geom_diagnostics(diagnostics = diagnostics,
table_theme = gridExtra::ttheme_default(base_size = 6),
ymin = 115, ymax = 130, xmin = 2010,
message = FALSE)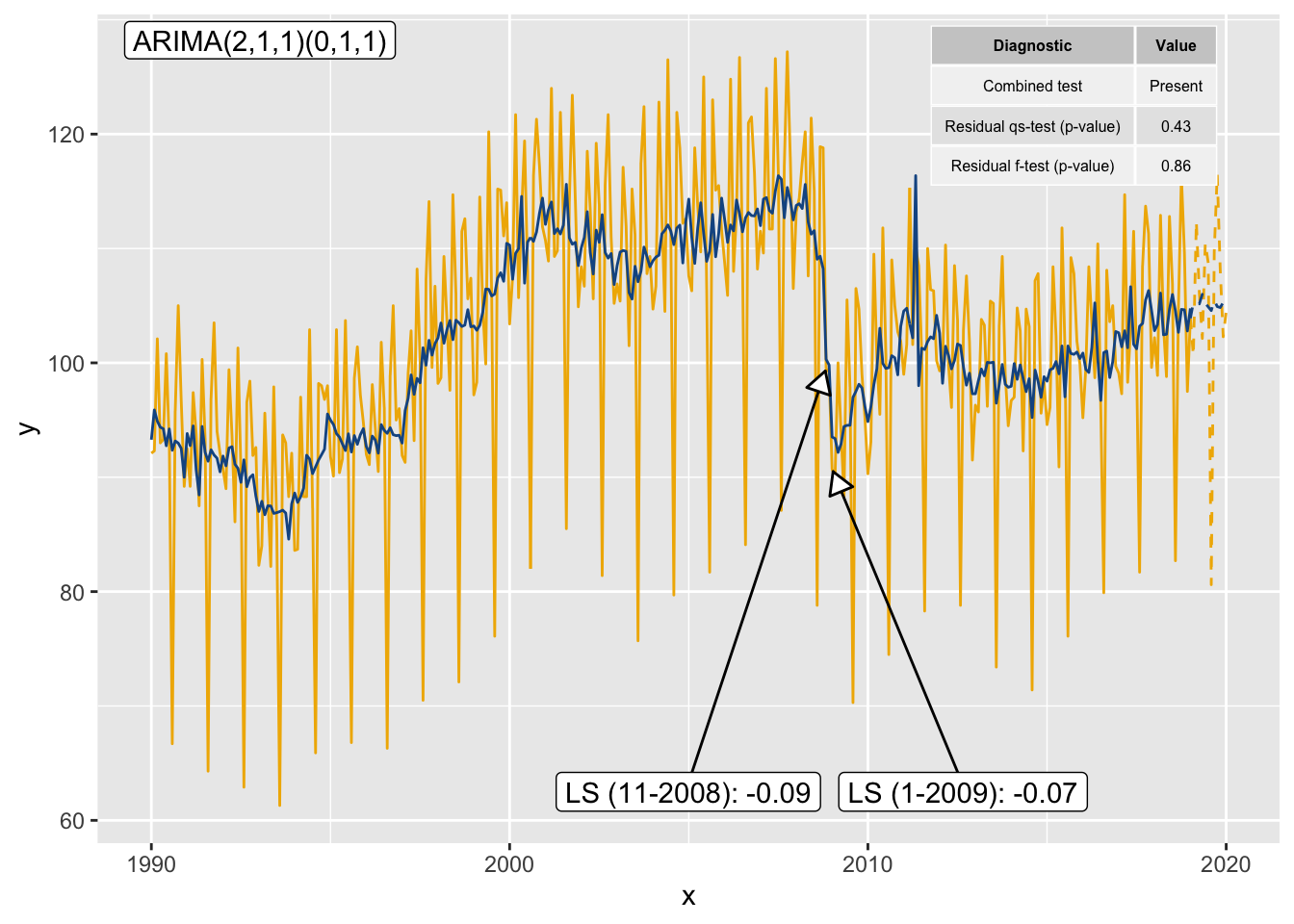
ggdemetra.
Pour combiner plusieurs graphiques on peut également utiliser le package patchwork :
library(patchwork)
p_diag <- init_ggplot(mod) +
geom_diagnostics(diagnostics = diagnostics,
table_theme = gridExtra::ttheme_default(base_size = 8),
message = FALSE) +
theme_void()
p_sa / p_diag +
plot_layout(heights = unit(c(4, 1.5), "null"))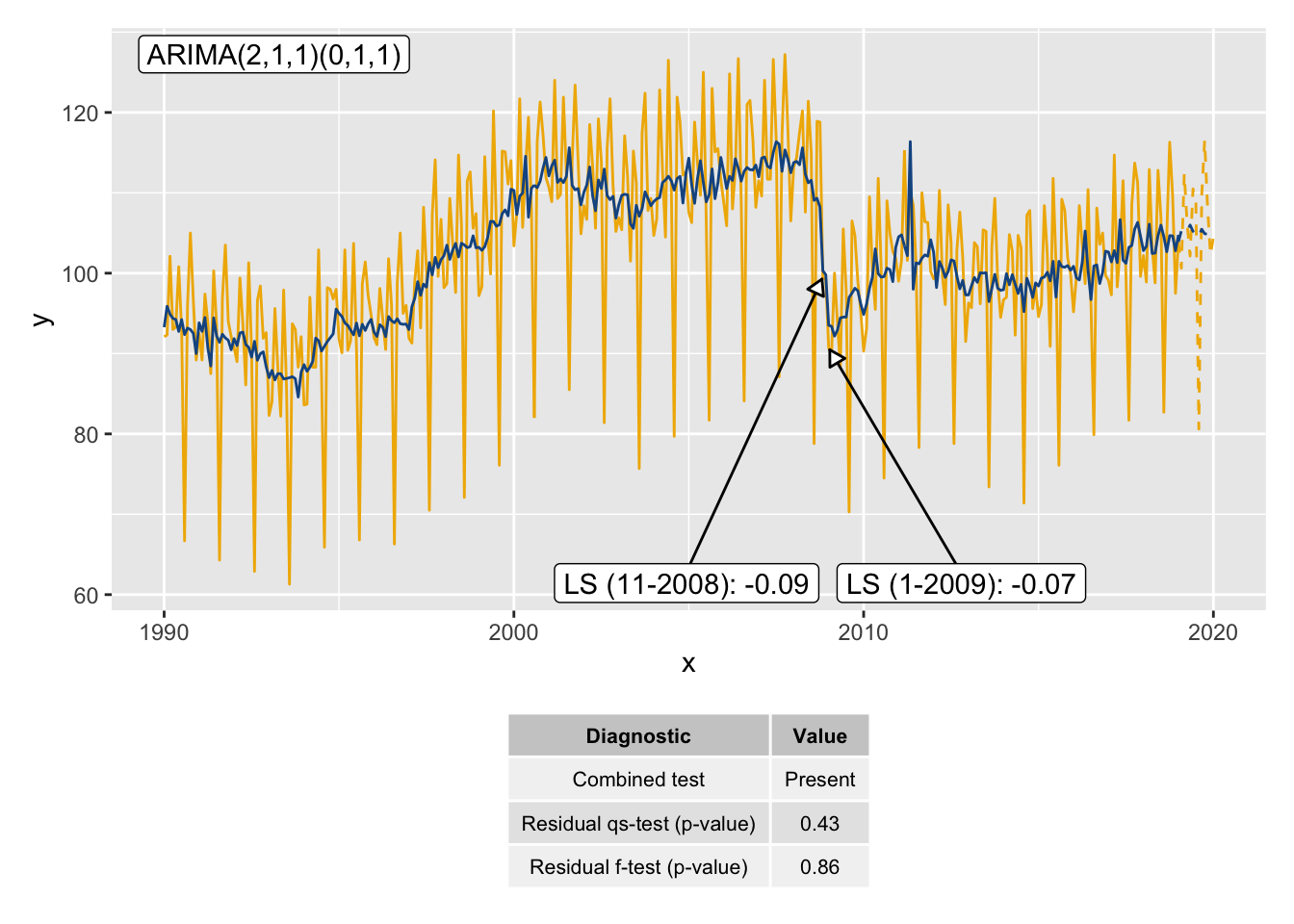
ggdemetra avec patchwork.
La fonction ggsiratioplot() permet de tracer les SI ratios avec ggplot2 :
ggsiratioplot(mod)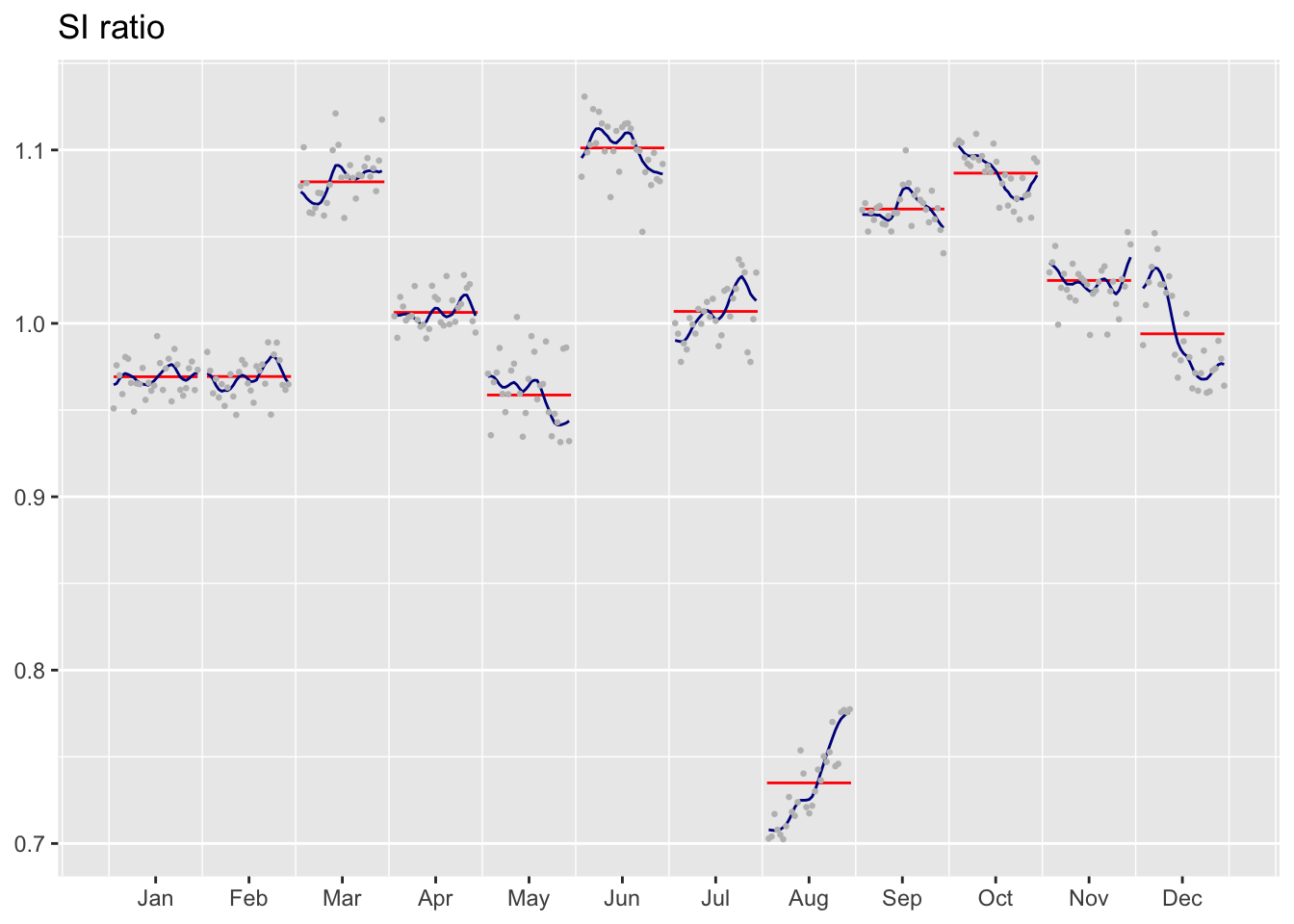
ggplot2 et ggdemetra::ggsiratioplot().
Les différentes composantes du modèle (ainsi que leurs prévisions) peuvent être facilement extraites via les fonctions calendar(), calendaradj(), irregular(), trendcycle(), seasonal(), seasonaladj(), trendcycle() and raw().
Créer une fonction graph_synthetique() qui prend en paramètre un modèle de RJDemetra et qui fait deux graphiques :
à gauche les séries brutes, désaisonnalisée et tendance sur les 10 dernières années ;
à droite les SI-Ratio.
Pour récupérer les 10 dernières années on pourra récupérer la dernière date connue avec la fonction time() et utiliser la fonction ggplot2::coord_cartesian() (à préférer à la fonction ggplot2::xlim() qui supprime des données et refait donc une désaisonnalisation sur une autre période).
graph_synthetique <- function (x, titre = NULL) {
y <- raw(x)
last_date <- time(y)[length(y)]
p_sa <- init_ggplot(x) +
geom_line(color = "#F0B400") +
geom_sa(component = "sa", color = "#155692") +
geom_sa(component = "t", color = "#1E6C0B") +
coord_cartesian(xlim = c(last_date - 10, NA)) +
# On change l'affichage de l'axe des abscisses
scale_x_continuous(labels = zoo::as.yearmon)
p_siratio <- ggsiratioplot(x,
# supprime le titre
main = NULL,
start = last_date - 10)
((p_sa + p_siratio) &
# on applique la fonction theme_bw à tous les graphiques
theme_bw()) +
# On ajoute un titre global
plot_annotation(
title = titre
)
}
graph_synthetique(mod)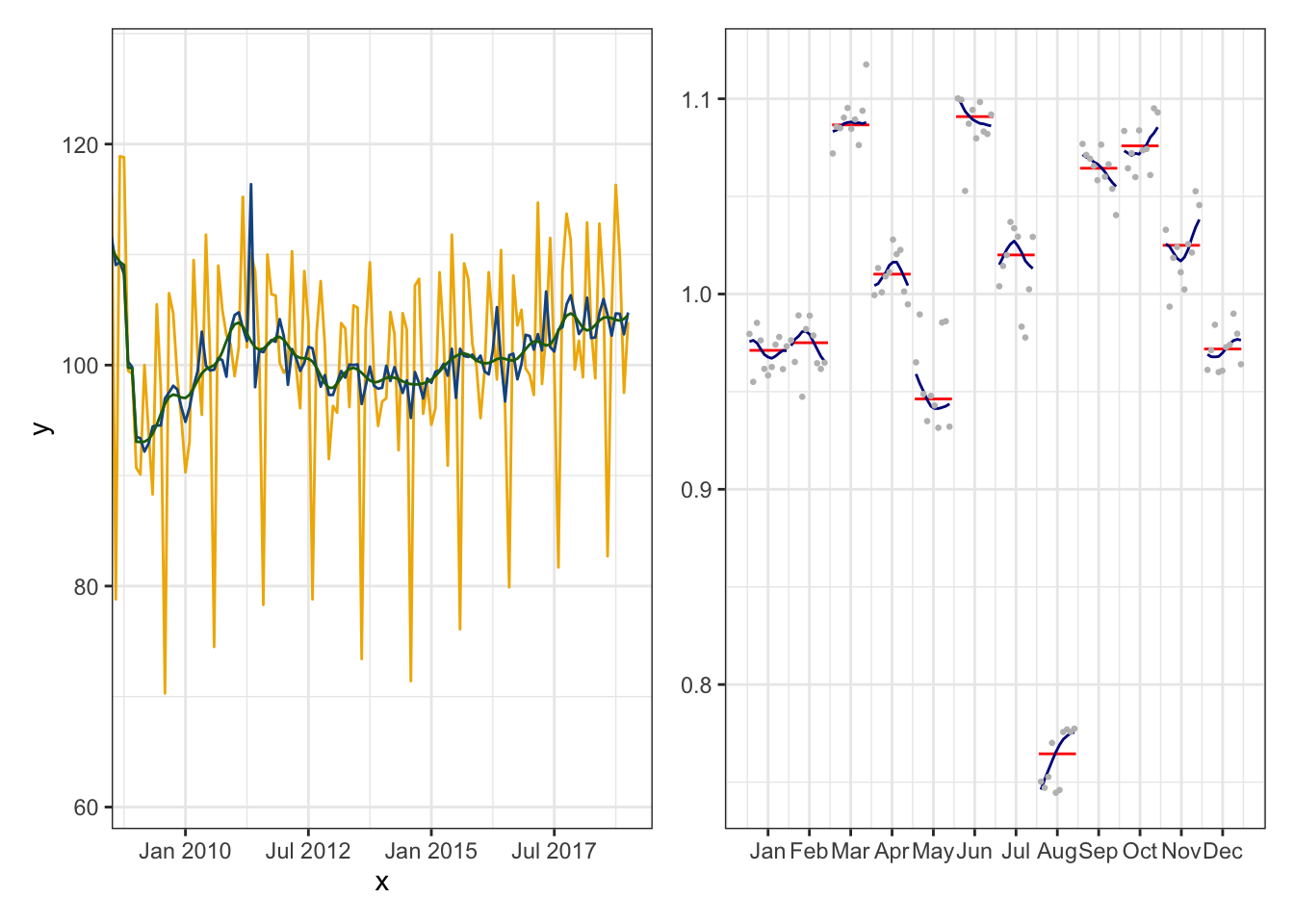
ggdemetra.
Ci-dessous un exemple de comparaison entre ggplot2::xlim() et ggplot2::coord_cartesian() :
p_cc <- init_ggplot(mod) +
geom_sa(component = "sa", color = "#155692") +
geom_sa(component = "t", color = "#1E6C0B") +
coord_cartesian(xlim = c(2010, NA), ylim = c(95, 120))
p_xlim <- init_ggplot(mod) +
geom_sa(component = "sa", color = "#155692") +
geom_sa(component = "t", color = "#1E6C0B") +
xlim(c(2010, NA)) +
coord_cartesian(ylim = c(95, 120))
p_cc / p_xlimWarning: Removed 240 rows containing non-finite outside the scale range
(`stat_sa()`).Frequency used: 12Warning: Removed 240 rows containing non-finite outside the scale range
(`stat_sa()`).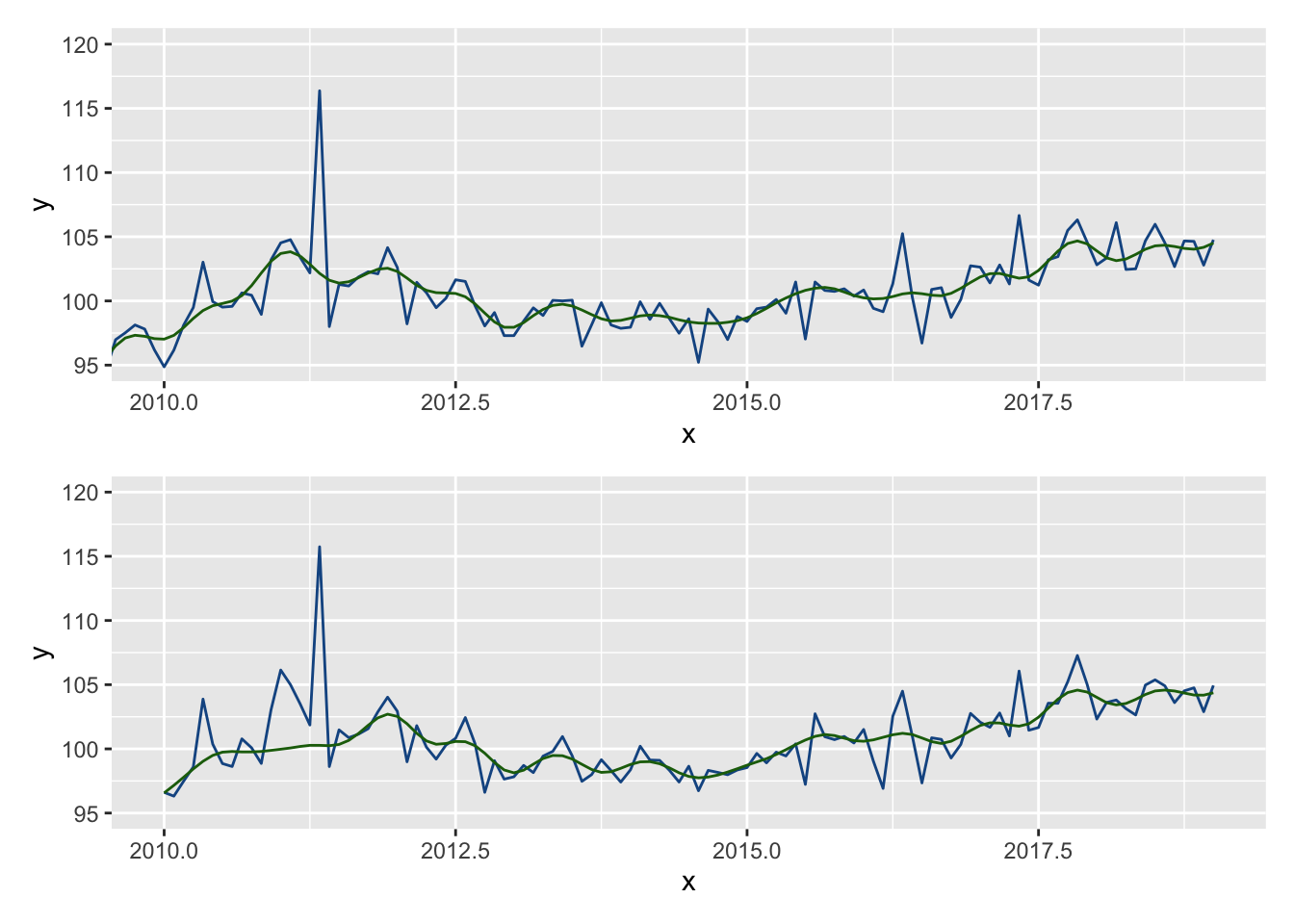
ggplot2::xlim() et ggplot2::coord_cartesian().
Un message et deux warnings sont générés : c’est parce que les données avant 2010 sont supprimées avec la fonction xlim() et qu’une nouvelle désaisonnalisation est alors effectuée (avec une spécification par défaut qui peut être différente de celle utilisée pour créer le modèle). Cela explique les différences entre les deux graphiques
À partir de la fonction précédente, on peut par exemple faire une fonction qui va lire un workspace et faire un cahier pdf le graphique précédent pour tous les modèles de chaque multiprocessing :
# Construisons d'abord un workspace exemple :
y <- ipi_c_eu[, "FR"]
jws <- new_workspace()
new_multiprocessing(jws, "X-13")
new_multiprocessing(jws, "TRAMO-SEATS")
for (spec in c("RSA5c", "RSA0", "RSA1", "RSA2c", "RSA3", "RSA4c")) {
add_sa_item(jws, "X-13", jx13(y, spec), spec)
}
for (spec in c("RSAfull", "RSA0", "RSA1", "RSA2", "RSA3", "RSA4", "RSA5")) {
add_sa_item(jws, "TRAMO-SEATS", jtramoseats(y, spec), spec)
}
dir <- tempdir()
save_workspace(jws, file.path(dir, "workspace.xml"))
jws <- load_workspace(file.path(dir, "workspace.xml"))
compute(jws)
all_m <- get_model(jws, progress_bar = FALSE)
all_m <- lapply(all_m, function(x){
# On enlève les espaces à la fin des noms,
# cela arrive souvent avec les fichiers Excel
names(x) <- gsub(" *$", "", names(x))
x
})
# Booléen pour supprimer les fichiers existants
replace_existing_file <- TRUE
# dossier contenant tous les graphiques
dir_exp <- "graph"
if (!dir.exists(dir_exp))
dir.create(dir_exp)
for (sap in names(all_m)) {
if (!dir.exists(file.path(dir_exp, sap)))
dir.create(file.path(dir_exp, sap))
for(series in names(all_m[[sap]])) {
# Si le fichier existe déjà on ne fait pas l'export
file <- file.path(
dir_exp, sap, paste0(
# on ne prend que les 20 premiers caractères pour les noms des fichiers
# (vite trop long si on part de fichiers Excel)
substr(series, 1, 20),
".pdf"))
if (!replace_existing_file && file.exists(file))
next;
print(sprintf("%s : %s", sap, series))
p_dashboard <- graph_synthetique(x = all_m[[sap]][[series]],
titre = series)
ggsave(filename = file, plot = p_dashboard,
# format A4 paysage
width = 29.7, height = 21, units = "cm")
}
}
# Ensuite on va créer un cahier avec tous les pdfs
for (sap in names(all_m)) {
qpdf::pdf_combine(input = sprintf("%s/%s/%s.pdf", dir_exp, sap,
substr(names(all_m[[sap]]), 1, 20)),
output = sprintf("%s/%s.pdf", dir_exp, sap))
}5.2 rjdqa
rjdqa permet de reproduire deux tableaux de bord différents (mais avec base R plutôt que ggplot2 pour des raisons de performance) :
sc_dashboard()qui reproduit le tableau de bord de Statistique Canada : voir notamment chapitre 22 du Handbook sur la désaisonnalisation https://ec.europa.eu/eurostat/web/products-manuals-and-guidelines/-/KS-GQ-18-001 ;simple_dashboard()qui reproduit un tableau de bord simplifié.
Si vous avez d’autres idées n’hésitez pas à contribuer au package ou à faire une issue sur github !
library(rjdqa)
mod <- tramoseats(window(ipi_c_eu[,"FR"], end = 2019))
plot(sc_dashboard(mod))
plot(simple_dashboard(mod))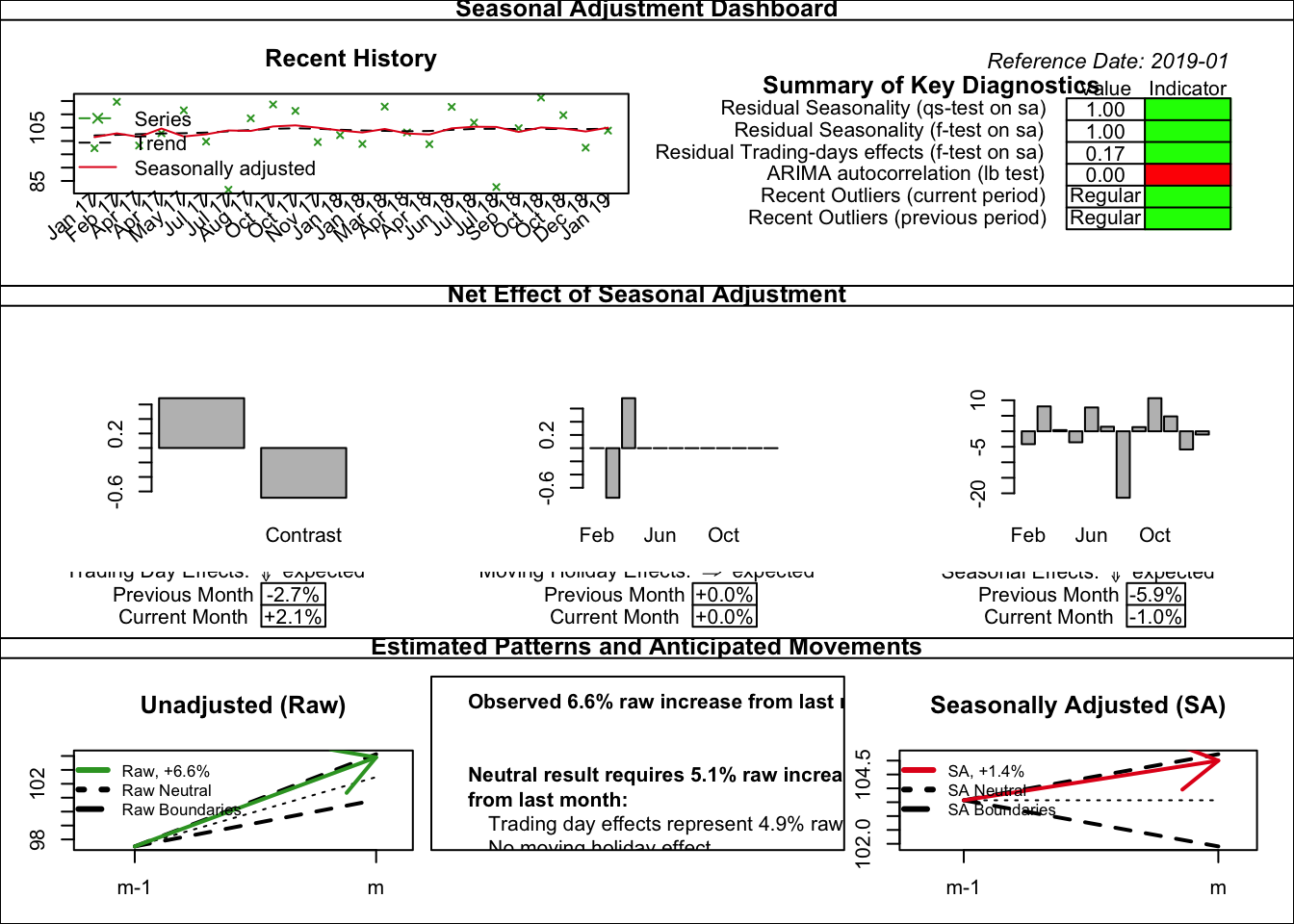
sc_dashboard().
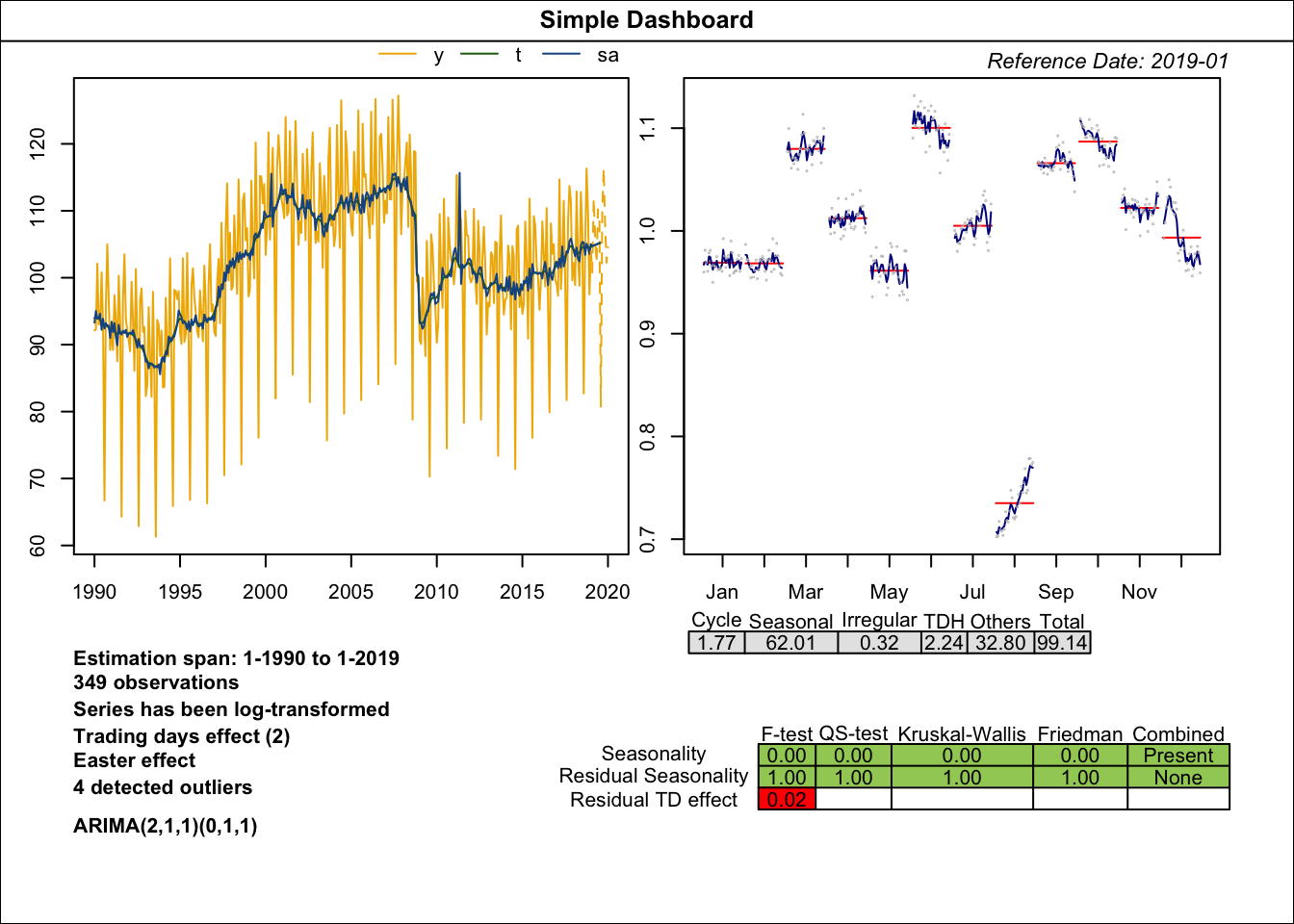
simple_dashboard().
5.3 rjdmarkdown
rjdmarkdown fournit différentes fonctions pour améliorer le rendu des modèles avec rmarkdown. Voir :
https://aqlt.github.io/rjdmarkdown/articles/rjdmarkdown-pdf.pdf pour le rendu en PDF ;
https://aqlt.github.io/rjdmarkdown/articles/rjdmarkdown-html.html pour le rendu HTML.
Un document peut également être généré de manière automatique à partir d’un workspace. Par exemple :
# # On peut reprendre le workspace précédent
# y <- ipi_c_eu[, "FR"]
# jws <- new_workspace()
# new_multiprocessing(jws, "X-13")
# new_multiprocessing(jws, "TRAMO-SEATS")
# for (spec in c("RSA5c", "RSA0", "RSA1", "RSA2c", "RSA3", "RSA4c")) {
# add_sa_item(jws, "X-13", jx13(y, spec), spec)
# }
# for (spec in c("RSAfull", "RSA0", "RSA1", "RSA2", "RSA3", "RSA4", "RSA5")) {
# add_sa_item(jws, "TRAMO-SEATS", jtramoseats(y, spec), spec)
# }
# dir <- tempdir()
# save_workspace(jws, file.path(dir, "workspace.xml"))
library(rjdmarkdown)
output_file <- tempfile(fileext = ".Rmd")
create_rmd(jws, output_file,
output_format = c("html_document", "pdf_document"),
output_options = list(toc = TRUE,
number_sections = TRUE))
browseURL(sub(".Rmd",".pdf", output_file, fixed = TRUE))
browseURL(sub(".Rmd",".html", output_file, fixed = TRUE))En réutilisant la fonction graph_synthetique() précédente :
output_file <- tempfile(fileext = ".Rmd")
out <- create_rmd(jws, output_file,
output_format = c("pdf_document"),
preprocessing_fun = NULL,
decomposition_fun = graph_synthetique,
diagnostics_fun = NULL,
output_options = list(toc = TRUE,
number_sections = TRUE),
knitr_chunk_opts = list(fig.pos = "h", echo = FALSE,
fig.cap = "Tableau de bord"))
browseURL(out)Notes de bas de page
Une autre façon de faire est d’importer le workspace
jws <- load_workspace("workspace.xml")et de le sauvegarder sans rien fairesave_workspace("workspace.xml"). L’export va renommer les noms des fichiers des multi-processings en utilisant leur nom, ce qui évitera cette opération de renommage.↩︎